| CATEGORII DOCUMENTE |
| Astronomie | Biofizica | Biologie | Botanica | Carti | Chimie | Copii |
| Educatie civica | Fabule ghicitori | Fizica | Gramatica | Joc | Literatura romana | Logica |
| Matematica | Poezii | Psihologie psihiatrie | Sociologie |
Functiile materialului genetic
Replicarea ADN
Replicarea ADN reprezinta transmiterea fidela a informatiei genetice la celulele fiice, in urma diviziunii celulare. Replicarea este una dintre cele doua functii esentiale ale materialului genetic. Mai mult decat atat, replicarea ADN este procesul de baza al continuitatii vietii pe Pamant.
1.1 Principalele etape ale replicarii ADN
Ca si alte procese din biologia moleculara, si procesul de replicare se desfasoara in 3 etape - initierea, elongarea si terminarea :
Initierea implica recunoasterea regiunii de pe o molecula ADN unde va incepe procesul de replicare
Elongarea cuprinde evenimetele ce se desfasoara la o bifurcatie de replicare, unde catenele parentale sunt copiate an catene fiice
Terminarea este o etapa destul de putin cunoscuta si se desfasoara atunci cand molecula parenatla a fost complet replicata
Etapele chimice ale replicarii ADN
Din punct de vedere chimic, replicarea ADN presupune urmatoarele etape principale:
desfacerea initiala a dublului helix intr-o zona denumita regiune de origine a replicarii
sinteza unor fragmente de ARN m.c. scurt ce ofera capatul 3'-OH liber pentru sinteza primelor legaturi fosfo-diesterice; aceste fragmente poarta numele de primeri si sunt sintetizate de ARN polimeraze speciale denumite primaze
in bucla de ADN desfacut procesul de replicare se va desfasura in ambele directii, formandu-se deci 2 bifurcatii de replicare
in fata fiecarei bifurcatii de replicare dublul helix va fi desfacut prin interventia topoizomerazelor (care derasucesc dublul helix) si a helicazelor (care desfac legaturile de hidrogen dintre cele 2 catene); astfel, bucla de replicare se va largi in fiecare din cele 2 capete
fiecare din cele 2 catene ale helixului parental va servi drept matrita pentru sinteza unei catene noi
sinteza catenelor noi se realizeaza de catre enzime numite ADN polimeraze, pe baza de complementaritate cu catena veche folosita ca matrita; nucleotidele sunt legate intre ele prin formarea de legaturi fosfo-diesterice; alungirea catenei noi se realizeaza in directie 5' - 3'
datorita faptul ca ADN polimerazele nu pot sintetiza o catena noua decat in directie 5' - 3', la fiecare bifurcatie de replicare, sinteza celor 2 catene noi are loc oarecum diferit :
una din catenele noi este sintetizata continuu, pornind de la un singur primer: aceasta catena este denumita catena conducatoare ("leading") (Figura 1)
cealalta catena este sintetizata discontinuu, pe masura ce dublul helix parental este desfacut in continuare; aceasta catena este denumita catena intarziata ("lagging") si este formata din multe fragmente distincte, denumite fragmente Okazaki (de la numele celul care le-a descris prima oara) de aproximativ 100 nucleotide; fiecare asemenea fragment este initiat de la un primer
intr-o faza mai avansata a replicarii primerii sunt indepartati atat din structura catenei conducatoare, cat si din cea intirziata, dupa care golurile sunt umplute tot de o ADN polimeraza
aceste fragmente sunt apoi unite intre ele prin refacerea legaturilor fosfo-diesterice de catre o ADN ligaza
in timpul procesului de replicare monocatenele ADN (atat cele vechi, cat si cele nou sintetizate) sunt stabilizate si protejate de actiunea nucleazelor, de catre o clasa speciala de proteine numite proteine Ssb (Single Stranded Binding)
terminarea replicarii unei molecule de ADN difera intre moleculele circulare si cele lineare
la moleculele circulare, intalnite in majoritatea cazurilor la cromozomul bacterian si la plasmide, cele 2 bifurcatii de replicare se intalnesc intr-o regiune denumita ter.
la moleculele ADN lineare din cromozomii de la eucariote, capetele acestora (telomerele) sunt replicate printr-un mecanism diferit, iar fragmentele sunt apoi unite de ADN ligaze
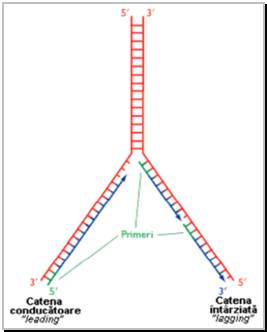
Figura 1 Reprezentarea schematizata a unei bifurcatii de replicare.
Complementaritatea dintre bazele azotate de pe cele 2 catene ale moleculei de ADN determina o structura perfect adaptata functiei de replicare, fiecare dintre cele 2 catene servind drept matrita pentru sinteza unei catene complementare.
In majoritatea cazurilor, replicarea se realizeaza pe model semiconservativ, macromoleculele de ADN fiice fiind formate dintr-o catena veche si una noua (Figura 2).
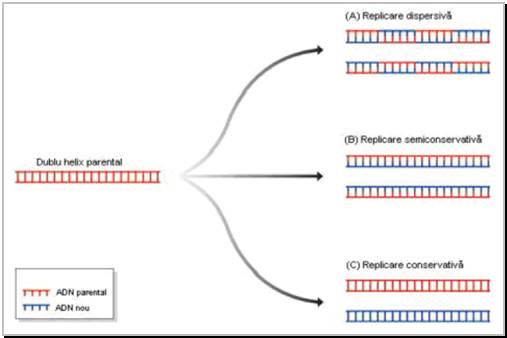
Figura 2 Principalele trei modele de replicare a moleculelor ADN.
La majoritatea organismelor (chiar si la genomuri virale) replicarea ADN se desfasoara bidirectional, ceea ce presupune formarea a doua bifurcatii de replicare ce avanseaza de la secventa de origine a replicarii (denumita secventa oriC pentru cromozomul bacterian) catre regiunea de terminare a replicarii (denumita secvnta ter in cromozomul bacterian).
Desi procesul de replicare a moleculelor de ADN se desfasoara enzimatic in mod similar la toate organismele, atat la cele procariote, cat si la cele eucariote, totusi exista suficiente diferente intre cele 2 tipuri principale de organisme.
Astfel, cromozomul bacterian este un replicon unic, adica are o singura secventa de origine a replicarii. In contrast, cromozomii de la eucariote sunt fiecare dintre ei structuri multirepliconice (au mai multe regiuni de origine a replicarii ADN) (Figurile 3 si 4).
|
|
|
|
|
Figura 3 Replicarea bidirectionala a unui cromozom bacterian (A) si a unui cromozom de tip eucariot (B) |
Figura 4 Reprezentarea schematica a replicarii cromozomului bacterian. |
Mai mult chiar, apar diferente si datorate structurii tertiare. Cromozomul bacterian are o structura circulara si, ca urmare, cele 2 bifurcatii de replicare se intalnesc la nivelul secventei ter (Figura 5). Cromozomul de tip eucariot are o structura lineara, iar capetele lui (denumite telomere) se replica diferit de restul cromozomului.
|
|
Figura 5. Imaginea de microscopie electronica si reprezentarea schematizata a unei molecule circulare de ADN in timpul replicarii. |
1.2 Enzimologia replicarii cromozomului bacterian
Cercetarile asupra procesului de replicare a moleculei ADN precum si asupra enzimelor implicate au debutat pe cromozomul de Escherichia coli si, ca urmare, majoritatea datelor experimentale provin de la bacterii (Figura 6).
1.2.1 Complexul primozom
Complexul primozom este format dintr-o serie de proteine ce determina initierea replicarii ADN. Cele mai importante proteine sunt:
primaza: la bacterii este denumita DnaG si este codificata de gena dnaG; aceasta enzima sintetizeaza scurte fragmente de ARN ce au functie de primeri, adica ofera capul 3'-OH liber pentru polimerizare.
proteinele de initiere: n', n, n", DnaA, DnaB, DnaC si DnaT
Initierea replicarii presupune detasarea secventei oriC de membrana plasmatica, dupa ce, in prealabil, a fost atinsa o anumita masa celulara (denumita masa de initiere) si in urma acumularii unei cantitati substantiale de fosfolipide acide pe fata interna a membranei plasmatice.
Secventa oriC din cromozomul de E.coli are 245 bp si prezinta 2 regiuni importante:
o regiune cu 4 "cutii" dnaA (sunt formate din cate 9 bp, denumite 9-meri) la care se va atasa proteina DnaA
o regiune cu 3 "cutii" de tip 13-meri, bogate in adenina-timina, la care se vor atasa complexe proteice DnaB-DnaC
Zece pana la 12 molecule de DnaA se leaga la "cutiile" dnaA, determinand buclarea moleculei de ADN si, ca atare, desfacerea dublului helix in "cutiile" dnaB. Zonele monocatenare aparute se complexeaza cu proteina DnaB (cu functie de helicaza), adusa de proteina DnaC. Ulterior, primaza (DnaG) incepe aici sinteza moleculelor de ARN primeri (Figura 7).
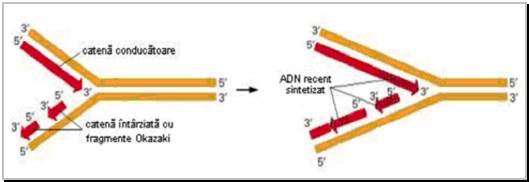
Figura 6 Reprezentarea schematica a unei bifurcatii de replicare.
|
|
Figura 7 Originea replicarii in cromozomul de E. coli. |
|
Figura 8 Structura regiunii de origine a replicarii la drojdia de bere (Saccharomyces cerevisiae). (A) Structura secventei ARS1, o secventa ARS tipica de la S.cerevisiae, cu pozitionarea secventelor functionale A, B1, B2 si B3. (B) Desfacerea dublului helix are loc in regiunea B2 prin atasarea proteinei ABF1 la regiunea B3. Proteinele complexului de origine a replicarii sunt atasate permanent la regiunile A si B1. |
|
In procesul de replicare a ADN, in diverse etape, apar zone monocatenare ce sunt stabilizate (si "aparate") fata de actiunea nucleazelor prin atasare de proteine de tip Ssb (Single Stranded Binding - proteine ce se ataseaza la monocatene ADN).
1.2.2 Topoizomerazele
Superspiralizarea cromzomului bacterian impune interventia unor enzime care sa realizeze relaxarea dublului helix ADN. Aceste enzime se numesc topoizomeraze si actioneaza asupra topoizomerilor ADN (Figura 9). Topoizomerii sunt molecule ADN cu structura primara si secundara identica si care difera in structura tertiara.
Doua molecule ADN d.c. ce au aceeasi secventa de nuceotide dar numar diferit de inlantuiri si suporarasuciri sunt topoizomere datorita diferentelor de natura topologica.
La bacterii, ca de altfel si la eucariote, exista 2 clase de topoizomeraze cu mecanisme diferite de actiune (Figura 9) :
topoizomeraze tip I - induc rupturi monocatenare (si tranzitorii) in ADN
topoizomeraze tip II - induc rupturi bicatenare in ADN (Figura 10)
La bacterii, topoizomeraza de tip I cea mai bine caracterizata este TopA de la E.coli, care relaxeaza suprarasuciri negative inalt rasucite. Topoizomerazele de tip II, in absenta ATP, au rolul de a relaxa macromolecula ADN ca si TopA, in timp ce in prezenta ATP induc suprarasuciri negative in moleculele circular covalent inchise (CCC) relaxate. Cea mai cunoscuta enzima din aceasta clasa este ADN giraza, care poate introduce aproximativ 100 de suprarasuciri negative per minut.
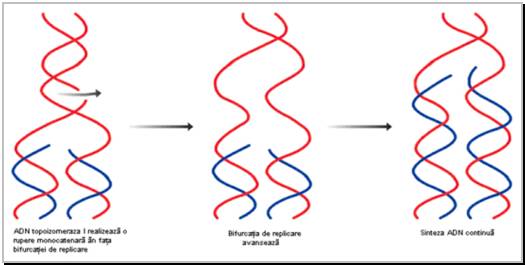
Figura 9 Desfacerea dublului helix intr-o bifurcatie de replicare, prin actiunea topoizomerazelor.
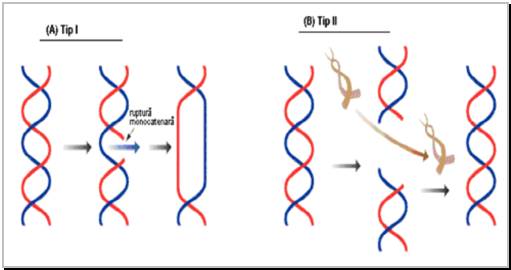
Figura 10 Modul de actiune al ADN topoizomerazelor de tip I si II.
(A) Topoizomerazele de tip I realizeaza ruperi monocatenare, conduc catena intacta prin aceasta rupere, refacand apoi legatura fosfo-diesterica. (B) Topoizomerazele de tip II taie ambele catene dintr-un dublu helix; prin acest spatiu este trecut un alt fragment al dublului helix, iar apoi se refac cele 2 legaturi fosfo-diesterice.
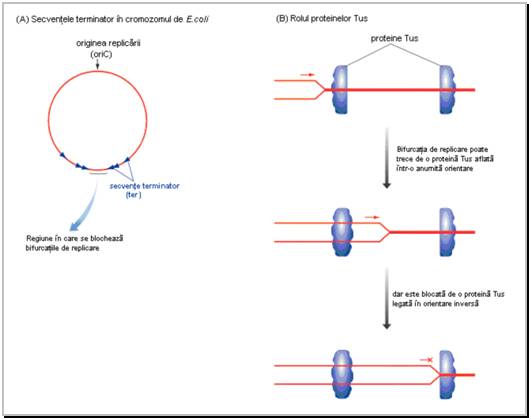
Figura 11 Rolul secventelor terminator si al proteinelor Tus in replicarea cromozomului la E.coli.
1.2.3 ADN polimeraze
Procesul de replicare propriu-zisa a ADN este realizat de un complex de enzime denumite ADN polimeraze. Cea mai importanta activitate enzimatica desfasurata de o asemenea enzima este formarea de legaturi fosfo-diesterice intre nucleotide adiacente, cu cresterea lantului in directie 5' - 3'.
Toate ADN polimerazele descrise pana in prezent polimerizeaza in directie 5' - 3' si nu pot realiza acest proces decat pornind de la capul 3'OH liber al unei nucleotide. De obicei, acest cap este oferit de un scurt fragment monocatenar de ARN denumit primer.
La bacterii au fost descrise 3 tipuri de ADN polimeraze ce coexista in aceeasi celula :
ADN polimeraza I (ADN pol I) este codificata de gena polA si are o g.m. de aprox 100 Md. In celula de E.coli exista circa 400 molecule de ADN pol I care are o viteza de polimerizare de 667 nucleotide / min. In afara de activitatea de polimerizare, aceasta enzima poate desfasura si activitate de exonucleaza (adica de desfacere de legaturi fosfo-diesterice si eliminare de nucleotide dintr-o catena). Astfel, ADN pol I are un domeniu polipeptidic ce realizeaza activitate exonucleazica in directie 3' - 5' , activitate foarte importanta ce ii asigura enzimei si posibilitatea de a isi corecta singura eventualele erori de incorporare a unor nucleotide imperecheate gresit. Aceasta functie este denumita "citire corecta" ("proof-reading"). Totodata, ADN pol I poate desfasura si activitate exonucleazica in directie 5' - 3', activitate ce ii permite indepartarea primerilor ARN.
ADN polimeraza II (ADN pol II) este codificata de gena polB. Si aceasta enzima poate avea si activitate exonucleazica 5' - 3'.
ADN polimeraza III (ADN pol III) este principala replicaza a cromozomului bacterian si reprezinta un adevarat complex enzimatic format din cel putin 10 subunitati functionale (Tabelul din Figura 12).
Figura 12 Subunitatile ADN polimerazei III de la procariote.
|
Denumirea subunitatii |
Masa moleculara |
Gena |
Functii |
Subansamblu |
|
130 |
pol C (dnaC) |
ADN pol Exo 5' - 3' |
Miezul enzimei |
|
|
27 |
dnaQ |
Exo 3' - 5' |
||
|
q |
10 |
holE |
structural |
|
|
t |
71 |
dnaXZ |
Dimerizarea miezului enzimei |
complexul t |
|
47 |
dnaXZ |
ATPaze |
complexul favorizeaza transferul sub. la matrita ADN |
|
|
35 |
holA |
|||
|
33 |
holB |
|||
|
c |
15 |
holC |
||
|
j |
12 |
holD |
||
|
40 |
dnaN |
Leaga miezul enzimei la ADN si il recicleaza |
complexul |
Holoenzima ADN pol III actioneaza ca dimer, fiecare din cei 2 monomeri realizand sinteza pe una din cele 2 catene. O exceptie o reprezinta complexul care actioneaza doar pe catena intarziata.
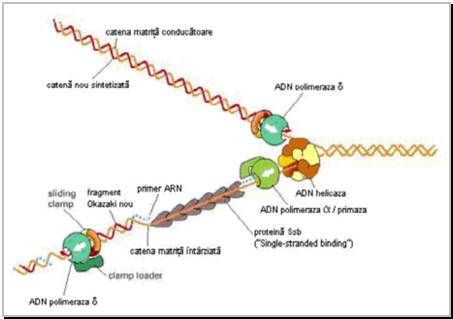
Figura 13 Reprezentarea schematizata a interventiei subunitatilor ADN polimerazei la eucariote.
1.3 Replicarea plasmidelor bacteriene
Indiferent daca confera bacteriei gazda caractere esentiale sau neesentiale, toate plasmidele au in structura lor o regiune foarte importanta pentru replicarea si mentinerea stabila a plasmidului respectiv in populatia bacteriana. In prezent se considera ca aceasta regiune esentiala ar contine:
a) secvente de origine a replicarii plasmidiale, denumite conventional oriV (fata de oriC - originea replicarii cromozomului bacterian); o secventa oriV trebuie sa indeplineasca urmatoarele conditii:
- regiunea ADN minimala, cu activitate in cis, ce poate sustine replicarea plasmidiala
- regiunea ADN in care cele 2 catene se despart pentru a initia procesul de replicare
- nucleotidul/nucleotidele la care incepe sinteza catenei leading
b) multe plasmide codifica o proteina implicata in initierea replicarii plasmidiale, denumita proteina de tip Rep
c) gene implicate in controlul replicarii plasmidiale
1.3.1 Replicarea plasmidelor lineare (ADN d.c. linear)
In ceea ce priveste replicarea plasmidelor lineare, aceasta se desfasoara prin mecanisme diferite functie de tipul de telomere.
a) plasmide cu telomere hairpin
Datorita capetelor legate covalent, ADN polimeraza poate trece peste ele continuand replicarea printr-un intermediar replicativ concatemeric. Asemenea plasmide ar putea reprezenta un caz special de plasmide circulare monocatenare, in care jumatate din cerc este complementara cu cealalta jumatate.
b) plasmide cu telomere invertroni
se replica printr-un mecanism de replicare ADN primata de proteine, in cazul de fata proteina TP (Telomeric Protein). In cazul unor asemenea plasmide, telomerele reprezinta originea replicarii si sunt recunoscute de proteine de initiere care desfac dublul helix ADN. Plasmidele cu telomere invertroni sunt replicate de o ADN polimeraza ce seamana mai mult cu ADN polimeraza a de la eucariote, decat cu ADN polimerazele de la procariote.
Ultima nucleotida de la capul 5' al fiecarei din cele 2 catene (nucleotida la care este atasata covalent proteina TP), este o adenina (A). Separat de molecula plasmidiala, se formeaza complexe din alt monomer TP si alta molecula de adenina. Cei 2 monomeri TP (unul atasat la o adenina, iar celalalt atasat la adenina din telomera plasmidiala) se atrag si are loc procesul de dimerizare, iar intre A si primul nucleotid (T) de pe catena complementara se formeaza legaturi de hidrogen. Tototdata, acest al doilea A ofera capul 3' liber pentru primarea polimerizarii (Figura 14).
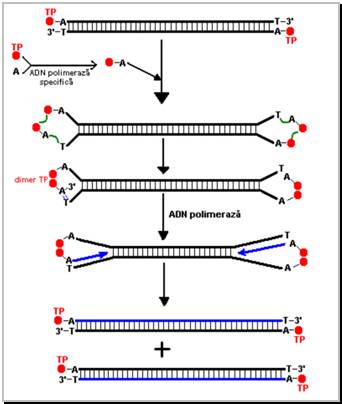
Figura 14 Replicarea plasmidelor lineare cu telomere invertroni.
1.3.2 Replicarea plasmidelor circulare.
Au fost descrise 3 mecanisme generale de replicare a plasmidelor circulare .
Acest mecanism prezinta asemanari foarte mari cu procesul de replicare a cromozomului bacterian. Ca si in cazul acestuia, acest mecanism poarta numele de "theta" datorita faptului ca intermediarii de replicare sunt molecule cu forma literei grecesti theta. Mecanismul theta de replicare se intalneste cu precadere la plasmidele de la bacteriile Gram-negative, dar si la unele plasmide de la Gram-pozitive (streptococ, enterococ, unii lactococi).
Replicarea acestor plasmide necesita o proteina initiator, codificata plasmidial si denumita proteina Rep, iar procesul porneste de la o regiune denumita oriV, ce are organizare similara cu oriC din cromozomul bacterian.
Intreg procesul de replicare de tip theta depinde de mai multe clase de proteine, din care insa doar proteinele de tip Rep sunt codificate plasmidial, celelalte sunt codificate cromozomal si sunt aceleasi proteine ce intervin si in replicarea cromozomului bacterian. O exceptie o reprezinta plasmidul Col E1, care se replica printr-un mecanism de tip theta, dar nu necesita prezenta unei proteine initiator de tip Rep.
La plasmidele ce se replica prin mecanism theta, regiunea oriV cuprinde: 4-5 secvente ADN in repetitie directa si denumite iteroni, la care se ataseaza proteina Rep (iteronii exista si in cazul celorlalte doua mecanisme de replicare plasmidiala), o regiune bogata in A/T, unde este initiata desfacerea dublului helix, cutii dnaA, la care se ataseaza proteina DnaA.
Proteinele Rep sunt proteine de tip leucine-zipper cu un domeniu HTH ("-helix-turnhelix") si se ataseaza la ADN intr-o maniera situs-specifica, si anume la secventele iteroni. Atasarea se realizeaza astfel incat moleculele Rep sunt aliniate pe aceeasi fata a moleculei ADN.
Etape
Procesul de replicare a plasmidelor prin mecanism theta se desfasoara in urmatoarele etape :
a) in prima etapa are loc atasarea proteinelor Rep la secventele iteroni; acest lucru determina o curbare a moleculei ADN in aceasta zona, fapt ce faciliteaza etapa urmatoare;
b) atasarea proteinelor DnaA la cutiile dnaA; la randul ei, aceasta etapa o faciliteaza pe urmatoarea;
c) atasarea complexului proteic DnaB-DnaC la regiunea din oriV bogata in A/T; DnaB este o helicaza si desface dublul helix in aceasta zona;
d) are loc sinteza unui ARN primer de catre o ARN polimeraza codificata de o gena cromozomala;
e) incepe procesul de polimerizare desfasurat, la majoritatea plasmidelor din aceasta clasa, de catre ADN polimeraza III (exceptie face plasmidul Col E1, care este replicat prin mecanism theta, dar de catre ADN polimeraza I);
f) sinteza celor 2 catene (leading si lagging) este cuplata si se desfasoara cvasi-simultan;
g) sinteza se desfasoara in majoritatatea cazurilor unidirectional, spre deosebire de replicarea cromozomului bacterian care la marea majoritate a speciilor bacteriene se desfasoara bidirectional.
Dintre cele mai cunoscute plasmide ce se replica prin mecanism theta, amintim pSC101, P1, RK2, RP4, R6K, R1, CColE2, ColE3.
1.3.2.2 Mecanismul Strand-Displacement (SD)
Denumirea provine din termenul strand-displacement (dislocarea catenei) si se refera la faptul ca in timpul procesului de replicare are loc dislocarea uneia din cele 2 catene ADN. Acest mecanism se intalneste mai ales la plasmidele promiscue din grupul de incompatibilitate IncQ. La aceste plasmide regiunea oriV este alcatuita din 3 secvente ADN de tip iteroni, 1 regiune bogata in G/C (174 bp), 1 regiune bogata in A/T (31 bp)
Procesul de replicare ADN de tip strand-displacement nu depinde de proteine ale celulei "gazda" (codificate cromozomal, cum ar fi DnaA, DnaB, DnaC), ci de trei proteine codificate plasmidial:
RepA - este o helicaza ce intra in dublul helix ADN in regiunea bogata in A/T si faciliteaza dislocarea catenei parentale nereplicate sub forma buclei D (forma literei D);
RepB - este o primaza care sintetizeaza un primer ARN necesar initierii polimerizarii ADN;
RepC - este o proteina initiator care se ataseaza la secventele iteroni din oriV.
Replicarea porneste de la doua origini (ssiA si ssiB) simetrice si adiacente, ce functioneaza in forma monocatenara si sunt situate pe cele 2 catene. Procesul de replicare incepe cand aceste 2 origini sunt expuse monocatenar.
Etape
Procesul de replicare SD se desfasoara in urmatoarele etape:
monomeri ai proteinei RepC se ataseaza la secvente iteroni din oriV;
proteina RepA (helicaza) se ataseaza la regiunea din oriV bogata in A/T, determinand desfacerea dublului helix;
proteina RepB (primaza) se ataseaza la bucla A/T si sintetizeaza un primer ARN;
incepe procesul de polimerizare ADN;
helicaza RepA faciliteaza dislocarea catenei parentale nereplicate sub forma unei bucle D.
Produsele intermediare ale unui proces de replicare SD sunt:
o molecula ADN circulara d.c.
o molecula ADN circulara m.c. - pe aceasta va fi initiat un nou proces de replicare care va conduce la formarea catenei complementare, si deci, a formei dublucatenare a plasmidului.
In cazul unui asemenea mecanism de replicare sinteza celor doua catene noi ale moleculei de ADN nu este cuplata si simultana.
1.3.2.3 Mecanismul Rolling-circle (RC)
Denumirea provine din faptul ca in timpul procesului, molecula de ADN are forma unui cerc rotativ. Acest mecanism de replicare este intalnit la plasmide din multe bacterii Gram-pozitive, de exemplu plasmidul pT181 (de la Staphylococcus), pUB110 si pC194 (de la Bacillus subtilis).
Ca si in cazul mecanismului SD, replicarea de tip RC depinde de o proteina initiator codificata plasmidial (propteina Rep) si de o serie de proteine codificate pe cromozomul bacterian.
Plasmidele ce se replica prin mecanism RC au doua secvente ori situate distantat una de alta:
dso ("double-stranded origin"), de la care este initiata replicarea primei catene noi (catena leading); dso are 2 regiuni: regiunea "bind", la care are loc atasarea proteinei initiator Rep si regiunea "nick", in care proteina Rep produce ruptura monocatenara initiala
sso ("single-stranded origin"), de la care este initiata sinteza celei de-a doua catene noi (catena lagging)
Etape
In procesul de replicare RC se parcurg urmatoarele etape:
proteina Rep se ataseaza la regiunea "bind" a secventei dso;
proteina Rep taie o legatura fosfodiesterica (ruptura monocatenara) si ramane atasata la capul 5' Tyr-fosfodiesterica; capul 3' va servi ca primer pentru sinteza catenei leading;
in procesul de sinteza a catenei leading intervin o serie de proteine codificate pe cromozomul bacterian: helicaza, Ssb, ADN polimeraza III;
replicarea catenei leading continua mai mult de o runda completa depasind regiunea dso;
pentru terminarea replicarii catenei leading are loc o reactie de transfer de catena: prin monomerul liber, proteina Rep ataca regiunea dso a catenei vechi, taie o legatura fosfodiesterica si reface alta, rezultand o molecula circulara d.c. care a eliberat si un dimer de Rep inactiv si, respectiv, o molecula circulara m.c. pe care, de la sso, este sintetizat un primer si apoi o catena noua
Deci, si in cazul acestui mecanism sinteza celor doua catene ADN noi nu este cuplata.
Intr-un asemenea proces proteina Rep are un rol mai complex decat intr-o replicare SD :
- se ataseaza la dso, avand rol in initierea replicarii;
- taie o legatura fosfodiesterica, producand nick-ul initial;
- la terminarea sintezei primei catene noi, Rep taie si reface legaturi fosfodiesterice
Datorita activitatii sale catalitice, proteina Rep de la plasmidele ce se replica printr-un mecanism RC este similara cu topoizomerazele din clasa I.
In general, plasmidele ce se replica prin RC au o alcatuire modulara, cel mai important modul fiind LIC ("Leading strand Initiation and Control region") care este format din dso, gena rep si elemente de control al replicarii plasmidei. Pe baza structurii LIC au fost definite 4 familii de plasmide ce se replica prin mecanism RC, familii ce au drept reprezentanti urmatoarele plasmide: pT181, pC194, pMV158, pSN2. Alte module importante in aceste plasmide sunt: 1-2 sso, 1 mob, 1 gena de antibiorezistenta.
1.3.3 Mecanisme de control al replicarii ADN plasmidial
Replicarea acestor plasmide se desfasoara unidirectional si este realizata de ADN polimeraza I si nu de ADN pol III care replica cromosomul bacterian.
Plasmidele din familia Col E1
Procesul de replicare incepe cu sinteza unui ARN primer (=ARN II), ce este transcris incepand de la un situs localizat la 555 pb in amonte fata de oriV. Initial, acest transcript este separat de ADN, ca in cazul unei transcrieri obisnuite. Pe masura ce ARN polimeraza se apropie de oriV, transcriptul incepe sa ramana hibridizat cu matrita ADN; cauza probabila: ARN II contine 6 G consecutive situate la circa 265 nucleotide in amonte fata de oriV; aceste 6 G interactioneaza probabil cu 6 C consecutive din ADN matrita, ce se gasesc la cca 20 nucleotide in amonte fata de oriV. Apoi hibridul este taiat de RNaza H, iar capul 3 al ARN va fi elongat de ADN pol I.
O modalitate de control negativ al replicarii plasmidelor din familia Col E1 este prin formarea unui al doilea ARN (=ARN I), care are functie de ARN antisens, contine 108 nucleotide si este transcris de la -445 fata de oriV, dar in directie opusa, de pe cealalta catena ADN.
Prin legarea lui ARN I (antisens) de ARN II (primer) este alterata conformatia secundara a acestuia din urma, astfel incat ARN primer nu mai poate hibridiza cu oriV.
Acest ARN antisens este transcris constitutiv, dar este instabil, iar legarea lui la ARN II este mediata de o proteina numita Rop (codificata de gena rop).
Aplicarea unui inhibitor al sintezei proteice (de exemplu, cloramfenicolul) blocheaza sinteza proteinei Rop, rezultatul fiind o crestere a frecventei de initiere a replicarii plasmidelor Col E1. Acest lucru se bazeaza pe faptul ca in citoplasma bacteriana exista suficiente molecule de ADN pol I (cca 300), chiar daca este blocata proteosinteza.
pSC101
Prima citare a lui pSC101 in literatura de specialitate a fost in 1973 - Cohen care a folosit-o ca vector in experimente de clonare. Ulterior insa, aceasta plasmida a fost identificata ca plasmida naturala la Salmonella si Escherichia coli. Ea prezinta un numar mediu de copii (6-7) per echivalent cromosomal si are o gena de rezistenta la tetraciclina. Intreaga plasmida (9263 pb) a fost complet secventiata.
Regiunea replicativa a acestei plasmide are 3 zone majore:
(1) regiunea par - foarte importanta pentru stabilitate si afecteaza si numarul de copii;
(2) regiunea ori - de la care porneste replicarea unidirectionala si care contine majoritatea situsurilor necesare pentru replicare si pentru incompatibilitate;
(3) gena repA - care este o gena specific plasmidiala si codifica o proteina de replicare.
pSC101, impreuna cu fagul P1 si cu o serie de alte plasmide (F, R6K), apartine unei clase de repliconi a caror replicare nu este realizata de ADN polimeraza I. Acesti repliconi codifica o proteina autoreglata (RepA) esentiala pentru replicare si poseda copii repetate direct ale unei secvente ce este specifica pentru fiecare plasmida si la care se ataseaza proteina RepA. RepA se ataseaza la cele 3 secvente RS din regiunea ori , participand la initierea replicarii.
|
|
|
|
Figura 15 Controlul replicarii la plasmidul Col E1. |
|
Mentinerea stabila a lui pSC101 in celula bacteriana presupune interventia a mai multor clase de proteine: DnaA (esentiala si in initierea replicarii cromosomului bacterian), IHF (Integration Host Factor, proteina histone-like implicata in reglarea unei serii de procese celulare), DnaB, DnaC, DnaG (primaza), componente ale holoenzimei ADN polimeraza III.
Regiunea ori din plasmidul pSC101 contine situsuri multiple de legare pentru proteine esentiale in replicarea plasmidiala:
- un situs de atasare a proteinei DnaA
- 2 secvente repetate, formate din cate 13 pb (13-mer), omoloage cu secventele din oriC si la care se ataseaza complexul DnaB-DnaC
- o regiune bogata in A/T
- intre regiunea A si regiunea T se gaseste situsul pentru IHF
- un cluster de 3 secvente repetate direct RS1 (24 pb), RS2 (18 pb), RS3 (24 pb) la care se atasaza proteina RepA
- ARN polimeraza se ataseaza la un promotor localizat in si intre aceste 3 secvente RS (promotorul pentru ARN-Y)
Procesul de transcriere in regiunea oriV si implicat in initierea replicarii este un fenomen extrem de frecvent in lumea bacteriana, atat pentru replicarea cromosomului, cat si a unor plasmide. In cazul lui pSC101, printr-un asemenea proces de transcriere la origine, este sintetizat ARN-Y care incepe din mijlocul lui RS2. Acest proces nu se afla sub controlul proteinei RepA, iar rolul moleculei ARN-Y pare sa fie acela de a facilita deschiderea dublului helix ADN in regiunea ori.
Regiunea par nu este esentiala pentru replicare (plasmidele par se replica), dar este esentiala pentru stabilitatea plasmidelor in celula bacteriana, probabil prin situsul pentru giraza.
Legarea simultana a proteinelor Rep A, IHF, Dna A, DnaB-Dna C este favorizata de curbarea ADN determinata de legarea IHF (curbeaza dublul helix ADN cu150o la situsul de atasare), si determina formarea replisomului.
Reglarea numarului de copii plasmidiale la pSC101 si, probabil, si la alte plasmide si sisteme similare, nu este produsul unui singur mecanism de reglare, ci este o rezultanta a unui set de interactiuni moleculare (ADN-proteine, ADN-ADN, proteine-proteine), fiecare dintre ele contribuind la replicarea, stabilitatea si partitia eficienta a plasmidului.
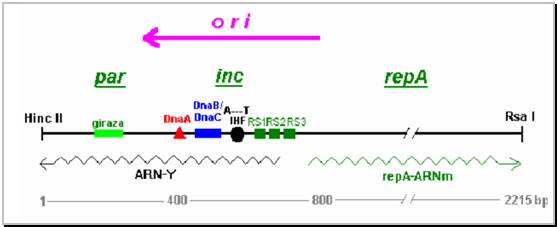
Figura 16 Structura regiunii replicative la plasmidul pSC101.
2 Transcrierea genetica
Principii si definitii
Transcrierea genetica reprezinta procesul de sinteza, catalizata enzimatic, a moleculelor de ARN, ca urmare a citirii informatiei codificate in molecule ADN. Procesul se desfasoara pe baza legilor de complementaritate chimica dintre cele 2 catene ale unei molecule de acid nucleic dublu catenar si conduce la formarea de legaturi fosfo-diesterice intre ribonucleotide.
Prin transcriere genetica se sintetizeaza toate tipurile de molecule ARN proprii celulelor, atat la organisme procariote, cat si la eucariote, si anume: ARN mesager (ARNm), ARN ribozomal (ARNr), ARN de transfer (ARNt), ARN heterogen nuclear (ARNhn).
Molecula ARN rezultata prin transcriere, inainte de orice alta procesare, poarta numele de transcript primar.
Procesul de transcriere a unei portiuni din ADN (denumita gena) presupune deci sinteza unei copii a informatiei genetice, copie care din punct de vedere chimic este o molecula de acid nucleic monocatenar, si anume ARN.
Transcrierea incepe in anumite zone din molecula ADN, zone numite promotori. Acestia au anumite secvente de nucleotide care ii fac usor de recunoscut de catre ARN polimeraze (enzimele ce sintetizeaza molecule de ARN). In zona promotorilor dublul helix ADN este desfacut de catre ARN polimeraze, formandu-se o asa-numita bucla de transcriere. In interiorul acesteia, ARN polimeraza sintetizeaza o molecula de ARN, copiind informatia genetica de pe una din catenele ADN pe care o foloseste ca matrita.
Primul nucleotid de pe catena ADN matrita care este citit si caruia ii corespunde un prim nucleotid in catena ARN, este numerotat conventional cu +1 si denumit startpoint (punct de pornire a transcrierii). Urmatoarele nucleotide din matrita ADN sunt numerotate +2, +3, +4 etc.
Fata de pozitia nucleotidului +1, se definesc 2 zone in matrita ADN :
- zona amonte (upstream), in care nucleotidele sunt numerotate cu semnul minus (-1, -2, -3 etc)
- zona aval (downstream), in care nucleotidele sunt numeortate cu semnul plus (+2, +3, +4 etc)
Procesul de transcriere pornit de la promotori continua pana in anumite zone din ADN, cu anumite secvente, zone denumite terminatori. In aceste zone, ADN polimeraza se desprinde de pe molecula de ADN, bucla de transcriere formata in dublul helix ADN se inchide, iar transcriptul ARN este eliberat.
O zona din ADN cuprinsa intre un promotor si un terminator poarta numele de unitate de transcriere. Din cele 2 catene ale moleculei de ADN, catena care este folosita ca matrita pentru sinteza unui transcript ARN este complementara cu aceasta si este numita catena antisens. Catena ne-matrita este denumita catena codificatoare sau catena sens.
Admitand ca o gena reprezinta o zona din ADN (sau mai exact, secventa de nucleotide de pe una din catenele ADN dintr-o anumita zona) ce codifica un polipeptid (sau o molecula de ARNr, ARNt ARNhn), o unitate de transcriere poate include :
- o singura gena, caz in care se numeste transcriere monocistronica
- sau mai multe gene, caz in care se numeste transcriere policistronica
Desi exista si destule exceptii, in general, transcrierea monocistronica este caracteristica organismelor eucariote, iar cea policistronica - procariotelor (Figura 16)
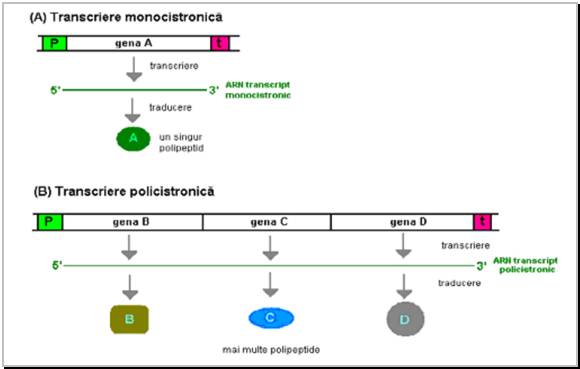
Figura 17 Reprezentarea schematizata a unui proces de transcriere monocistronica si, respectiv, policistronica.
2.2 Enzime
Procesul de transcriere genetica este catalizat de o clasa speciala de enzime numite ARN polimeraze.
La organismele procariote eixsta cate o singura specie moleculara de ARN polimeraza per celula, aceasta realizand sinteza tuturor tipurilor de ARN din celula.
La eucariote, majoritatea celulelor detin cate 3 specii moleculare de ARN polimeraze, fiecare dintre ele sintetizand anumite specii de ARN.
2.3 Transcrierea la procariote
2.3.1 Promotori
Promotorii reprezinta secvente din structura ADN, aflate in amonte fata de ogena, la care se ataseaza in mod situs-specific (in functie de secventa de nucleotide) ARN polimeraza.
Un promotor de la procariote prezinta 4 regiuni importante din punct de vedere functional :
- o secventa hexamerica (adica formata din 6 perechi de baze) in jurul pozitiei -35; este denumit generic hexamerul -35
- o secventa hexamerica in jurul pozitiei -10, denumita generic hexamerul -10
- o regiune spatiatoare intre cei 2 hexameri (ADN spacer)
- o regiune situata intre pozitiile -40 si -60, bogata in A si T, denumita elementul UP (Upstream = amonte)
Cei 2 hexameri si elementul UP au secventa de nucleotide inalt conservata, secventele consensus fiind 5'-TTGACA-3' pentru hexamerul -35 si, respectiv, 5'-TATAAT-3' pentru hexamerul -10 (Figura 18).
Cu cat secventele de nucleotide din cei 2 hexameri sunt mai apropiate de cele de mai sus, cu atat taria promotorului respectiv este mai mare, adica cu atat ARN polimeraza se va atasa mai strans si cu atat gena respectiva va fi transcrisa cu o rata mai ridicata.
Cei 2 hexameri din promotori sunt recunoscuti de subunitatea a ARN polimerazei, iar la elementul UP se ataseaza subunitatea -CTD a acestei enzime.
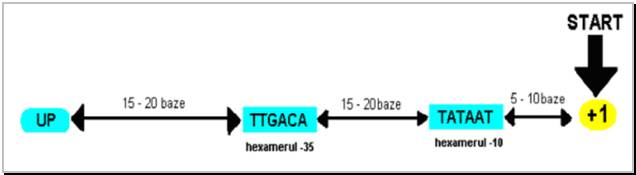
Figura 18 Structura promotorilor la procariote.
2.3.2 Terminatori
Transcrierea genetica continua de la promotori pana la terminatori. Acestia reprezinta regiuni din molecula ADN ce prezinta o anumita secventa de nucleotide :
- 2 copii ale unei secvente poli-GC in repetitie inversa; aceasta regiune prezinta complementaritate intracatenara si determina formarea in transcriptul ARN a unei structuri in ac-de-par (hairpin) ce impiedica avansarea ARN polimerazei
- o regiune formata din 4 - 10 adenine (ce corespunde pe transcript cu 4 - 10 resturi de uracil) care reprezinta semnalul propriu-zis de terminare a transcrierii
Desi regiunile terminator sunt identificate pe molecula ADN, terminarea transcrierii este de fapt realizata de catena ARN (Figura 19).
Pana in prezent au fost identificate 2 clase de terminatori la procariote :
- terminatori Rho - independenti : sunt regiuni de tip terminator in care cele 2 secvente poli-GC prezinta complementaritate intracatenara perfecta, realizand o structura in ac-de-par stabila
- terminatori Rho - dependenti : sunt regiuni terminator in care cele 2 poli-GC nu prezinta complementaritate intracatenara perfecta; in acest caz structura in ac-de-par este stabilizata de catre o proteina numita proteina Rho (de la litera greceasca Rho - r) care aluneca pe molecula ARN pana la acul-de-par si il stabilizeaza.
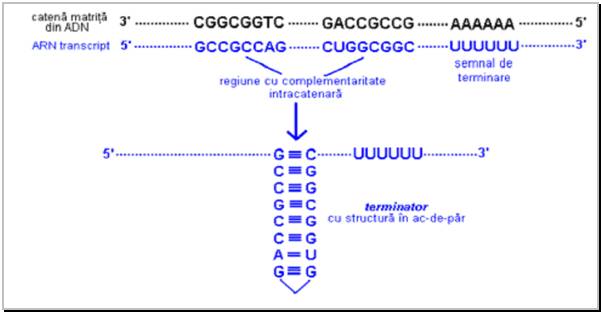
Figura 19 Structura terminatorilor la procariote.
2.3.3 ARN polimeraza de la procariote
Aceasta enzima este una dintre cele mai mari proteine din celula bacteriana, avand o greutatea de 480 kd, un diametru de aproximativ 100 Angstromi si fiind vizibila si la microscopul electronic. O celula de E.coli contine in medie 7000 de molecule de ARN polimeraza.
Holoenzima este formata din 4 tipuri de subunitati: , b, b' si Subunitatea este dimerizata, formul generala a enzimei fiind 2a - - ' - .
Subunitatea este codificata de gena rpoA si este dimerizata. Ea are un prim rol in asamblarea tuturor subunitatilor enzimei, proces ce se desfasoara in ordinea: Fiecare monomer de este format din 3 regiuni:
-CTD reprezinta domeniul carboxi-terminal al subunitatii si se ataseaza direct la ADN, recunoscand secventa UP din structura promotorilor.
-NTD reprezinta domeniul amino-terminal al subunitatii ; nu se ataseaza direct la ADN, ci la subunitatea
- linker polipeptidic ce are o structura flexibila si face legatura dintre cele 2 domenii terminale
Subunitatea este codificata de gena rpoB si realizeaza formarea propriu-zisa a legaturilor fosfo-diesterice intre ribonucleotide, acest proces desfasurandu-se intotdeauna in directie 5' - 3'.
Subunitatea este codificata de gena rpoC si are rol in atasarea initiala, situs-nespecifica a ARN polimerazei la molecula de ADN (Figura 20).
Subunitatea
Celula procariota contine mai multe specii moleculare de subunitate , fiecare recunoscand anumiti promotori si, deci, transcriind anumite gene. Astfel, in celula de E.coli, cele mai des intalnite specii moleculare de sunt:
are o g.m. de 70 kd si codificata de gena rpoD; acest recunoaste cele 2 secvente consensus TTGACA si TATAAT, fiind folosit de celula bacteriana in conditii generale de mediu. Ca urmare, marea majoritate a genelor bacteriene sunt transcrise cu ajutorul acestei subunitati
are g.m. 60 kd, este codificata de gena rpoN si este folosita de celula in conditii de privare de azot.
are g.m. de 32 kd, este codificata de gena rpoH si este folosita de celula in conditii de soc termic; cu ajutorul acestei subunitati sunt transcrise genele de soc termic.
are g.m. 24 kd, nu este cunoscuta gena codificatoare si este folosita de celula in conditii de soc termic extrem; se pare ca 24 participa la transcrierea unui set de gene ce determina o moarte celulara programata, o "sinucidere" celulara (asemanatoare proceselor de apoptoza de la celulele eucariote).
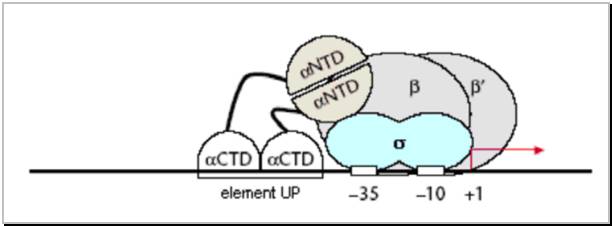
Figura 20 Reprezentarea schematica a atasarii ARN polimerazei procariote la promotori.
2.3.4 Fazele transcrierii genetice la procariote
Ca si alte procese realizate de materialul genetic, transcrierea genetica se desfasoara in 3 etape - initierea, elongarea si terminarea transcrierii.
Initierea transcrierii
Aceasta etapa incepe prin atasarea ARN polimerazei la un promotor, proces ce se desfasoara el insusi in mai multe etape:
a) etapa de promotor inchis I - ARN polimeraza se ataseaza la hexamerul -35
b) etapa de promotor inchis II (promotor curbat) - ARN polimeraza se ataseaza apoi si la hexamerul -10; cei 2 hexameri nu sunt insa orientati steric optim fata de situsurile de atasare la ARN polimeraza (si anume lla subunitatea s); ca urmare, pentru a se atasa la amandoi hexamerii, enzima trebuie sa distorsioneze molecula de ADN; deci, promotorul curbat prezinta o tensiune torsionala ce va fi eliberata prin deschiderea dublului helix pe o distanta de 10 - 18 pb, ajungandu-se astfel in cea de-a treia etapa, cea de
c) etapa de promotor deschis; se formeaza astfel bucla de transcriere care depaseste situsul START (nucleotidul +1 al catenei matrita)
Se formeaza astfel un complex binar: ADN - ARN polimeraza. Initierea transcrierii continua cu atasarea primului nucleotid, care de obicei la procariote este ATP sau GTP. Complexul devine astfel din binar, ternar: ADN - ARN polimeraza - ARN. Dupa sinteza a aproximativ 8-10 ribonucleotide, subunitatea se desprinde din complex, considerandu-se ca acest eveniment incheie etapa de initiere a transcrierii.
Elongarea transcrierii
Aceasta etapa incepe dupa desprinderea subunitatii si este realizata de subunitatea care formeaza legaturi fosfo-diesterice intre ribonucleotide. Catena ARN creste in directia 5' - 3'. Odata cu ARN polimeraza, si bucla de transcriere se deplaseaza pe molecula de ADN, transcriptul ramanand in hibrid cu matrita ADN doar pe o portiune de aproximativ 12 nucleotide.
Rata de polimerizare a ARN polimerazei de la E.coli este de circa 30 - 40 nucleotide per secunda. Frecventa erorilor de incorporare (a ribonucleotidelor gresit imperecheate cu cele din catena matrita ADN) de catre aceasta enzima este de 1 baza gresita per 104 baze incorporate. Dupa ce ARN polimeraza a trecut de promotor, o alta enzima (cu tot cu subunitate ) se poate atasa la acesta si poate incepe o noua runda de transcriere.
Terminarea transcrierii
Terminarea transcrierii are loc in momentul in care ARN polimeraza ajunge in regiunea unui terminator. Acul-de-par format in catena transcriptului (vezi figura 18) blocheaza avansarea enzimei si o determina sa stationeze cateva milisecunde; acest timp este insa suficient pentru ca transcriptul sa se desprinda din hibridul cu catena ADN matrita (desprinderea este facilitata si de faptul ca legaturile dintre poli-A de pe ADN si poli-U din ARN sunt slabe). Bucla de transcriere se inchide si astfel procesul este incheiat.
Moleculele de ARN transcript pot evolua fie ca ARN measger, ARN ribozomal, ARN de transfer.
Majoritatea moleculelor de ARNm de la procariote sunt monocistronice si sunt alatuie din 3 regiuni mai importante :
- regiunea cap (leader), care contine secventa SHINE-DALGARNO (5'-AGGAGG3') ce este complementara cu un hexamer de la capul 3' al moleculelor de ARNr de 16S
- regiunea codificatoare, care incepe cu codonul start AUG si se termina cu un codon stop
- regiunea cozii
2.4 Transcrierea la eucariote
2.1 ARN polimerazele la eucariote
In sine, procesul de transcriere la organismele eucariote se desfasoara in mod similar cu cel de la procariote. Exista totusi o serie de diferente.
Astfel, in timp ce la procariote exista o singura specie moleculara de ARN polimeraza per celula, la eucariote exista, in majoritatea cazurilor 3 specii moleculare de asemenea enzime. Cele 3 ARN polimeraze de la eucariote sunt inrudite si structural si functional. Cu toate acestea, cele 3 enzime initiaza transcrierea de la promotori diferiti si transcriu gene diferite :
- ARN polimeraza I - transcrie in mod special gene ce codifica pentru ARN ribozomal
- ARN polimeraza II - transcrie mai ales gene ce codifica ARN mesager
- ARN polimeraza III - transcrie mai ales ARN de transfer si o serie de molecule mici de ARN, de exemplu ARN nuclear mic si nucleolar mic
Alte diferente importante apar si in ceea ce priveste factorii de transcriere. Astfel, daca la procariote initierea transcrierii necesita doar facotrul , la eucariote debutul acestui proces necesita mai multe proteine, denumite generic factori de transcriere generali.
In general, structura promotorilor pentru ARN pol I si III este mai simpla decat a promotorilor pentru ARN pol II, desi si aceste 2 enzime necesita factori de transcriere.
2.2 Promotorul la eucariote
Regiunile promotor la eucariote prezinta o zona "miez" care cuprinde un set minimal de secvente necesare initierii transcrierii de catre ARN pol II. Un asemenea "miez" de promotor este de obicei format din 40 de nucleotide ce cuprind de multe ori si situsul START (nucleotidul +1) si este format din :
- elementul BRE ce reprezinta elementul de recunoastere a factorului de transcriere TFIIB (TFIIB Recognition Element)
- cutia TATA la care se ataseaza factorul TBP (TATA Binding Protein); aceasta proteina reprezinta de fapt o subunitate a factorului de transcriere TFIID
- regiunea Inr (Initiator) la care se ataseaza factorul TFIID
- regiunea DPE (Downstream Promoter Element = elementul in aval fata de promotor) la care se ataseaza tot TFIID
Cei mai multi promotori de la eucariote includ doar 2 sau 3 din aceste regiuni (Figura 21).
2.3 Complexul de pre-initiere
Factorii de transcriere necesari ARN polimerazei II au fost notati TFII (Transcription Factors for RNA polymerase II).
La eucariote se formeaza mai intai un complex preoteic de pre-initiere care se ataseaza la elementul TATA. Succesiunea de evenimente este urmatoarea :
- elementul TATA este recunoscut de TFIID; aceasta proteina este un complex cu mai multe subunitati, dintre care una (TBP) se va lega la cutia TATA.
- celelalte subunitati ale complexului TFIID poarta numele de proteine TAF (TBP Associated Factors = factori asociati cu TBP)
- TBP se leaga la cutia TATA; prin legarea sa la cutia TATA, TBP distorsioneaza molecula de ADN in acea regiune determinand "recrutarea" si a altor factori de transcriere si a ARN polimerazei
- TFIIA si TFIIB se ataseaza la promotor
- TFIIF se cupleaza cu ARN polimeraza si impreuna se ataseaza la promotor
- TFIIE si TFIIH se ataseaza si ei la intreg acest complex nucleo-proteic
Ca urmare a atasarii acestor proteine, dublul helix se deschide in zona promotorului. Spre deosebire de procariote, la eucariote deschiderea dublului helix necesita energie (furnizata prin hidroliza ATP) iar procesul este mediat de activitatea de tip helicaza a factorului TFIIH
Sinteza catenei ARN poate acum sa inceapa.
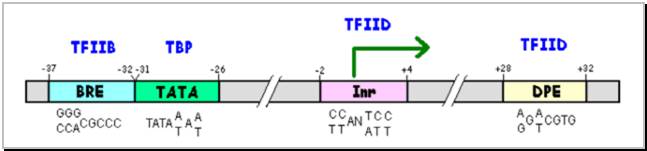
Figura 21 Structura unui promotor la eucariote.
Figura 22 Factorii generali de transcriere pentru ARN polimeraza II
|
Factori generali de transcriere |
Numarul de subunitati |
|
TBP |
1 |
|
TFIIA |
2 |
|
TFIIB |
1 |
|
TFIIE |
2 |
|
TFIIF |
3 |
|
TFIIH |
9 |
|
proteine TAF |
11 |

2.4 Factorii de elongare
In aceasta etapa, ARN polimeraza II se desprinde de marea majoritate a factorilor de initiere. Locul lor este luat de un set de factori de elongare (TFIIS, TEF). Astfel, TFIIS asigura icorporarea corecta a ribonucleotidelor si corecteaza bazele incorporate gresit (functie de "proofreading"). Factorul TEF fosforileaza anumite reziduuri de serina din structura ARN polimerazei II, fapt ce stimuleaza etapa de elongare.
Pe de alta parte, in timpul etapei de elongare, ARN polimeraza se asociaza cu o serie de proteine necesare pentru procesarea tipurilor de ARN transcript.
- enzime ce adauga la capul 5' al transcriptului un rest de guanina metilata, ceea ce ii va conferi transcriptului rezistenta la enzime de tip nucleaze si se ataseaza la structura ribozomului
- enzime ce produc poliadenilarea capatului 3': enzime de tip poli-A polimeraze adauga o coada de pana la 200 de resturi de A la capul 3' al transcriptului; atasarea unei asemenea enzime pare sa fie implicata in terminarea transcrierii
Bibliografie selectiva
Alberts B., Johnson A., Lewis J., Raff M., Roberts K., Walter P., 2002, Molecular Biology of the Cell. 4th ed., Garland Publishing House, New York.
Berg J.M., Tymoczko J.L., Stryer L., 2002, Biochemistry. , W. H. Freeman and Co., New York.
Brown T.A., 2002, Genomes. 2nd ed., BIOS Scientific Publishers, Ltd, Oxford, UK.
Campbell A.M., 1992, Chromosomal insertion sites for phages and plasmids. Journal of Bacteriology 174(23):7495-7499.
Cohen S.N., 1993, Bacterial plasmids: their extraordinary contribution to molecular genetics. Gene 135:67-76.
Cooper G.M., 2000, The Cell - A Molecular Approach. 2nd ed., Sinauer Associates, Inc., SUA.
Cornea C.P., 1998, Elemente de Inginerie Genetica, Editura ALL, Bucuresti.
Covic M., Stefanescu D., Sandovici I., 2004, Genetica medicala, Editura Polirom.
Dale J.W., 1998, Molecular Genetics of Bacteria, 3rd Edition.Anonymous Chichester, UK:John Wiley & Sons.
Del Solar G., Giraldo R., Ruiz-Echevarria M.J., Espinosa M., Diaz-Orejaz R., 1998, Replication and control of circular bacterial plasmids. Microbiol Molec Biol Rev 62(2):434-46
Embley T.M., Hirt R.P., Williams D.M., 1994, Biodiversity at the molecular level: the domains, kingdoms and phyla of life. Philos Trans R Soc Lond B Biol Sci 345(1311):21-33.
Espinosa M., del Solar G., Rojo F., Alonso J.C., 1995, Plasmid rolling circle replication and its control. FEMS Microbiol Lett 130:111-120.
Freifelder D., 1987, Microbial Genetics. Jones and Bartlett Publishers. Boston, USA.
Gavrila L., 2003, Genomica - Un tratat despre genom, de la virusuri la om, vol.I si vol.II, Editura Enciclopedica, Bucuresti.
Gilson E., Bachellier S., Perrin S., Perrin D., Grimont P.A.D., Grimont F., Hofnung M., 1990, Palindromic unit highly repetitive DNA sequences exhibit species specificity within Enterobateriaceae.. Researches in Microbiology 141:1103-1116.
Griffiths A.J.F., Miller J.H., Suzuki D.T., Lewontin R.C., Gelbart W.M., 1999, Introduction to Genetic Analysis. 7th ed., W. H. Freeman & Co., New York.
Hardy K.G., 1988, Plasmids: A Practical Approach. IRL Pres. Oxford, UK.
Herlea V., 1998 - Microbiologie generala, Ed. Univ., Bucuresti.
Hinnebusch H., Barbour A.G., 1992, Linear- and circular-plasmid copy numbers in Borrelia burgdorferi. J.Bact. 174(16):5251-5257.
Hinnebusch J., Tilly K., 1993, Linear plasmids and chromosomes in bacteria. Molecular Microbiology 10(5):917-922.
Hiraga S., 1992, Chromosome and plasmid partition in E.coli. Annu Rev Biochem 61:283-306.
Lewin B. , 1997, Genes. 6th ed., Oxford University Press, New York, USA.
Lodish H., Berk A., Zipursky S.L., Matsudaira P., Baltimore D., Darnell J.E., 2000, Molecular Cell Biology. 4th ed., W. H. Freeman & Co. Publishing, New York.
Manen D., Caro L., 1991, The replication of plasmid pSC101. Molecular Microbiology 5(2):233-237.
Margulis L., 1992, Biodiversity: molecular biological domains, symbiosis and kingdom origins. Biosystems 27(1):39-51.
Moller-Jensen J., Gerdes K.J.R., 2000, Plasmid and chromosome segregation in prokaryotes. Trends in Microbiology 8(7):313-320.
Nester E.W., Evans Roberts C., Nester M., 1995, Microbiology, A Human Perspective, WCB Publishers, SUA.
Pettijohn D.E., 1988, Histone-like proteins and bacterial chromosome structure. Journal of Biological Chemistry 263(26):12793-12796.
Pettijohn D.E., 1988, Histone-like proteins and bacterial chromosome structure. Journal of Biological Chemistry 263(26):12793-12796.
Prescott L.M., Harlez J.P., Klein D.A., 1996, Microbiology. Third Edition, WCB Publishers, SUA.
Raicu P., Stoian V., 1991, Genetica dezvoltarii la eucariote, Editura Academiei Romane.
Russel P.J., 1994, Fundamentals of Genetics, Harper Collins College Publishers, SUA.
Sakaguchi K., 1990, Invertrons, a class of structurally and functionally related genetic elements that includes linear DNA plasmids, transposable elements and genomes of adeno-type viruses.. Microbiological Reviews 54(1):66-7
Stoica I., Vassu T., Sasarman E., 2002 - Biologia si taxonomia moleculara a microorganismelor. Colectia de culturi microbiene. Ed. Arvin Press.
Strachan T., Read A.P., 1999, Human Molecular Genetics 2. 2nd ed., BIOS Scientific Publishers, Ltd,Oxford, UK.
Summers D.K., 1996, The Biology of Plasmids.Anonymous Anonymous Oxford, UK:Blackwell Science Ltd.
Sykora P., 1992, Macroevolution of plamids: a model for plasmid speciation. Journal of Theoretical Biology 159:53-65.
Trun N.J., 1998, Architecture of a bacterial chromosome. ASM News 64(5):276-283.
Vassu T., Stoica I., Cstuak O., Musat F., 2001, Genetica microorganismelor si Inginerie genetica microbiana. Note de curs si Tehnici de laborator. Editura Petrion, Bucuresti.
Watson J.D., Baker T.A., Bell S.P., Gann A., Levine M., Losick R., 2004, Molecular Biology of the Gene. Fifth Edition, CSHL Press.
Weaver R.F., 1999, Molecular Biology, WCB McGraw-Hill Press.
Woese C.R., Kandler O., Wheelis M.L. , 1990, Towards a natural system of organisms: proposal for the domains Archaea, Bacteria and Eucarya., Proc Natl Acad Sci USA, 87, pp 4576-4579.
Zarnea G., Tratat de Microbiologie Generala. vol I (1983), II (1984) si V (1994), Editura Academiei Romane.
|
Politica de confidentialitate | Termeni si conditii de utilizare |

Vizualizari: 6128
Importanta: ![]()
Termeni si conditii de utilizare | Contact
© SCRIGROUP 2025 . All rights reserved